中科院常兴研究组发现RNA剪接基因编辑的新方法
中科院常兴研究组发现RNA剪接基因编辑的新方法
2018年10月5日,国际知名学术期刊《分子细胞》在线发表了中国科学院上海生命科学研究院(营养与健康研究院)常兴研究组题为“Genetic modulation of RNA splicing with a CRISPR-guided cytidine deaminase”的最新研究成果。证明可以利用TAM (Targeted-AID induced mutagenesis)基因编辑,靶向DNA上的RNA剪接顺式元件,高效调控RNA剪接,用于研究RNA可变剪接的功能,以及用于人类遗传疾病的治疗。
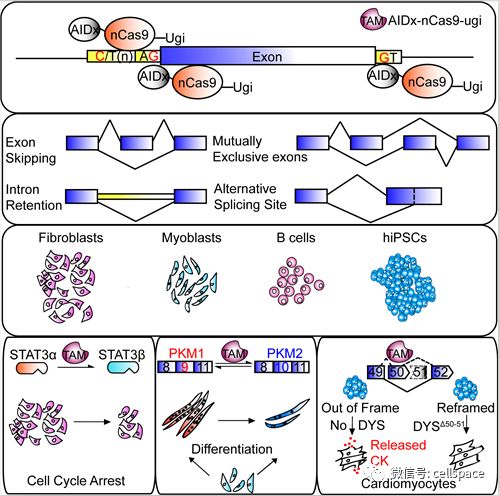
TAM 融合蛋白高效调控外显子跳读、互斥外显子选择、可变剪接位点选择和内含子包含
真核细胞中, RNA剪接是基因的表达的重要环节。据估计,超过75%的人类基因具有一种以上的mRNA剪接方式 (可变剪接),其中大部分可以翻译为功能性蛋白质。但相对于基因功能而言,对于可变剪接的生理功能,认识还非常有限,原因主要在于调控内源RNA剪接的实验手段非常有限。RNA剪接的异常也是许多疾病的直接诱因,估计35~50%的人类疾病由基因剪接异常造成。因此,无论是从学术研究或是临床应用的角度,都亟需开发出调控RNA剪接的基因编辑新方法。
常兴研究组过去开发TAM (Targeted AID-induced mutagenesis), 也就是融合核酸酶活性缺陷的Cas9蛋白和胞嘧啶脱氨酶AID,并且发现它具有两个特点。首先,可以在sgRNA靶向的DNA上,将C/G碱基随机向其它碱基突变,可以用于分析肿瘤耐药性突变及诱导蛋白体外进化等;其次,在偶联UNG抑制剂UGI后,可以在将sgRNA靶向区域中一个小窗口内(5-6个碱基)的C高效向T突变 (Nature Methods, 2016)。
在这一新研究中,博士研究生袁娟娟和马云青首先注意到,98%以上的内含子有保守的GU(内含子开始)和AG(内含子末尾)序列,推测如果可以高效精确的将G突变成A, 可以特异性阻断外显子识别,调控内源性mRNA的剪切。通过这一策略,利用TAM诱导剪接位点DNA上G>A突变,就可以诱导可变外显子(alternative exon)及组成性外显子(constitutive exon)的跳读 (Exon skipping);改变可变剪接位点的选择(Alternative splice site);调节互斥外显子的选择 (mutually exclusive exons);诱导小的内含子的包含 (intron retention)。此外,如果通过TAM将3’剪接位点上游polypyrimidine track中包含的C向T突变,可以促进下游外显子的包含。因此,利用TAM可以成功的实现针对RNA剪接的“loss of function”和”gain of function”调控。
最后,研究人员探索了利用TAM修复杜氏肌营养不良症(DMD)的可行性。DMD是一种致命的遗传疾病(发病率男性中1/4000)。其发病原因在于,遗传突变造成Dystrophin蛋白的完全缺失,引发肌肉萎缩和瘫痪,最终造成心脏或肺功能的衰竭。如果通过外显子跳读,能产生内部截短的Dystrophin蛋白,可以达到对DMD的治疗效果。因此研究人员构建了DMD病人来源的诱导型多能干细胞, 此病人因为外显子删除造成Dystrophin蛋白的完全缺失。通过在剪接位点诱导G>A的突变,研究人员实现了目标外显子的完全跳读,在所有表达TAM的细胞中恢复了Dystrophin的蛋白表达,修复了心肌细胞的缺陷。
该研究得到了科技部、国家自然科学基金委、中国科学院先导计划和上海市科委相关项目的资助。(科技处)
论文链接:https://www.sciencedirect.com/science/article/pii/S109727651830741X